先日定期健康診断があり、初めての胃カメラに挑戦。
そのデータをもらえたので、解析、jpeg画像に変換してみた。
というのも、検査終了後、医師と結果を見ながら説明してもらったのだが、自分でもその画像をよく見てみたいと思った。
自分でも滅多に見ることのできない自分の体の中の映像だ。ダメ元でもらえないか聞いてみたところ、千円程度の手数料さえ払えばCD-Rに焼いてくれるとのことだったので依頼。
通常はなにか検査結果に問題があったときに紹介状と共に渡されるものだと思う。
なお、毎年バリウムの検査を受けていたのだが、胃カメラのほうが確実な検査ができるらしく、たまにはやっておこうということで今回初めて受けてみることにした。(無事問題はなかったので一安心)
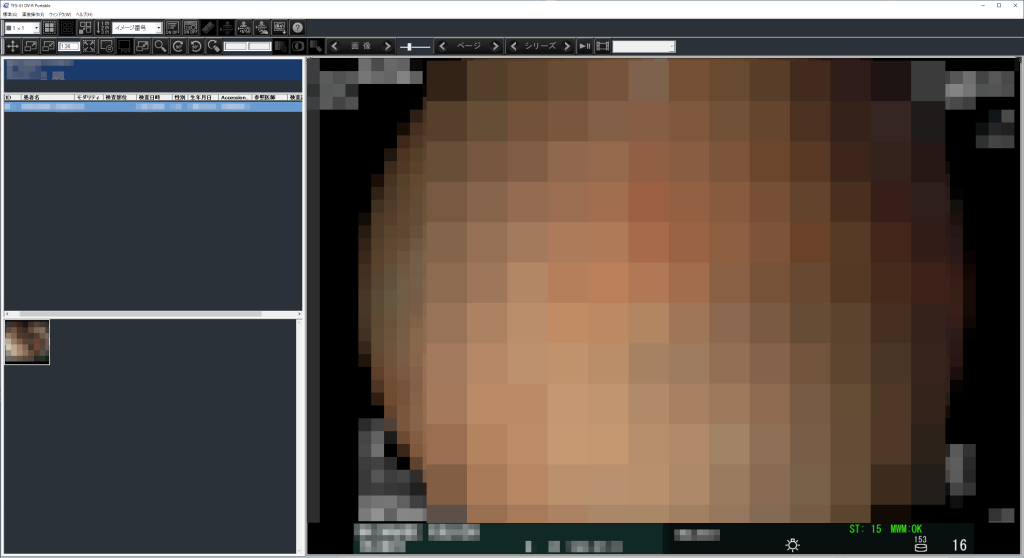
CD-Rに同梱されていたビューア
帰って早速、PCでCD-Rを挿入したところ、病院で見せてもらうような、画像閲覧用のアプリケーションが起動するようになっており、これで確認することができる。
(個人情報や画像にはモザイクかけてます)
生データの解析
が、やはり生の画像データとしても取り出したい。
単純に画像ファイルが並んでいることを期待してCD-Rのファイル構成を確認したところ、特に画像ファイルなどは見当たらない。DICOMというディレクトリの下に拡張子のない連番データが並んでいる。
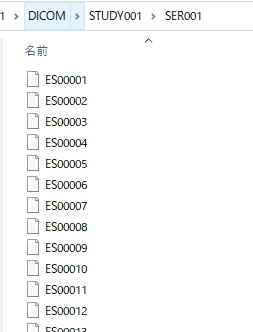
これが画像データだろう。jpegなどの画像データであれば拡張子をつけてやるだけで良いかもしれない。
まずはヘッダで判別するべくバイナリデータを見てみた。

すると先頭は0x57c, 0x1a0a58などあるのでググってみたがわからず。
しかし、その下の方にDICMという文字列がある。これを調べたところ、
医療用画像のDICOMというフォーマットらしいことがわかった。
医用画像の国際規格 DICOM
DICOM(ダイコムと読む)医用画像の国際規格とのこと。
ファイル名
- 半角大文字および数字
- 最大8文字
- 拡張子は存在しない
8文字+拡張子3文字のMS-DOSのファイル名規則より厳しく、かなり古いコンピュータとの互換性を想定したものらしい。
が、実際には.dcmなどの拡張子が一般的とのこと。
Mime-Typeもある
application/dicomというMime Typeも定義されているらしい。
ファイル構造
| ヘッダ | プリアンブル | 128バイト | 固定。先頭から。 通常は0x00でパディングすることになっており、読み飛ばせば良い |
| ヘッダ | プリフィックス | 4バイト | 固定。”DICM”という文字列。プリアンブルに続く。 DICOMファイルかどうかはここを見て判別すべきである。 |
| データセット | メタ情報、データ本体 | 可変長 | メタ情報と本体のデータセットが複数連なる。 |
プリアンブルは正確には任意に使ってよい領域とのことなので、独自フォーマットのデータの埋め込みもありそう。(なので上記のように何かしらの値が入っているケースに遭遇した)
データセットの構造
| オフセット | |
| 0-3 | タグ。データ本体が何の情報かをあらわす。 0-1:グループ、2-3:エレメント グループが偶数:標準タグ。DICOM規格に定義されている。 グループが奇数:プライベートタグ。独自の形式を設定できる。 |
| 4-5 | VR(Value Representation) データ本体の型をあらわす * 明示的VR, 暗黙的VRがある。後者はVRが存在しないがタグから判定可能。 |
| 6-11 | データ長(2-6バイト可変。VRから推定) |
| 12- | データ本体 |
参考
- https://medicalware.org/wiki/DICOM%E3%83%95%E3%82%A1%E3%82%A4%E3%83%AB
- http://docs.leadtools.grapecity.com/v19/dh/to/leadtools.topics.dicom~di.topics.dicomfilestructure.html
データを直接確認してみる
性質上、一般には使われないことから、フリーで使えるようなアプリケーションはあまりなさそう。
だが、国際規格ならgithubあたりにライブラリがあるだろうということで、pythonライブラリを見つけた。
メタ情報を見てみる
from pydicom import dcmread
import glob
l = glob.glob("./*.dcm") # 各DICOMファイルを*.dcmにリネームしておいた
for path in l:
print(path)
ds = dcmread(path)
print(ds)
成功。実装は簡単だが、こんな感じで結構色々な情報が含まれていることがわかる。
医用だけあって、検査日時や機器、患者の情報などが埋め込まれていることがわかる。(一部個人情報などは伏せている)
.\ES00001.dcm
Dataset.file_meta -------------------------------
(0002, 0000) File Meta Information Group Length UL: 212
(0002, 0001) File Meta Information Version OB: b'\x00\x01'
(0002, 0002) Media Storage SOP Class UID UI: VL Endoscopic Image Storage
(0002, 0003) Media Storage SOP Instance UID UI: 1.X.XXX.XXXXXX.XXXX.XXXXXXXXXXXXXXX.XXXXXXXXXXX.1
(0002, 0010) Transfer Syntax UID UI: Explicit VR Little Endian
(0002, 0012) Implementation Class UID UI: 1.2.392.200036.9116.7.28.1
(0002, 0013) Implementation Version Name SH: 'TMSCR_TFS01_1.0'
(0002, 0016) Source Application Entity Title AE: 'FMSES01'
-------------------------------------------------
(0008, 0005) Specific Character Set CS: ['', 'ISO 2022 IR 13', 'ISO 2022 IR 87']
(0008, 0008) Image Type CS: ['ORIGINAL', 'PRIMARY']
(0008, 0016) SOP Class UID UI: VL Endoscopic Image Storage
(0008, 0018) SOP Instance UID UI: 1.X.XXX.XXXXXX.XXXX.XXXXXXXXXXXXXXX.XXXXXXXXXXX.1
(0008, 0020) Study Date DA: '20210824'
(0008, 0021) Series Date DA: '20210824'
(0008, 0022) Acquisition Date DA: '20210824'
(0008, 0023) Content Date DA: '20210824'
(0008, 0030) Study Time TM: '104743'
(0008, 0031) Series Time TM: '104743'
(0008, 0032) Acquisition Time TM: '104919'
(0008, 0033) Content Time TM: '104920'
(0008, 0050) Accession Number SH: 'XXXXXXXXXXX'
(0008, 0060) Modality CS: 'ES'
(0008, 0070) Manufacturer LO: 'FUJIFILM Corporation'
(0008, 0080) Institution Name LO: 'XXXXXXXXX'
(0008, 0090) Referring Physician's Name PN: ''
(0008, 1010) Station Name SH: ''
(0008, 1030) Study Description LO: ''
(0008, 1040) Institutional Department Name LO: ''
(0008, 1050) Performing Physician's Name PN: ''
(0008, 1090) Manufacturer's Model Name LO: 'EP-6000'
(0008, 1111) Referenced Performed Procedure Step Sequence 0 item(s) ----
(0008, 2111) Derivation Description ST: ''
(0008, 2112) Source Image Sequence 0 item(s) ----
(0010, 0010) Patient's Name PN: 'XXXXXXXXX^XXXXXXXXX'
(0010, 0020) Patient ID LO: 'XXXXXXXXX'
(0010, 0021) Issuer of Patient ID LO: ''
(0010, 0030) Patient's Birth Date DA: 'XXXXXXXXX'
(0010, 0040) Patient's Sex CS: 'M'
(0010, 1010) Patient's Age AS: 'XXXXXXXXX'
(0010, 4000) Patient Comments LT: 'XXXXXXXXX'
(0018, 1000) Device Serial Number LO: 'XXXXXXXXX'
(0018, 1020) Software Versions LO: 'Ver2.470'
(0018, 1030) Protocol Name LO: 'General'
(0020, 000d) Study Instance UID UI: 1.X.XXX.XXXXXX.XXXX.XXXXXXXXXXXXXXX.XXXXXXXXXXX.1
(0020, 000e) Series Instance UID UI: 1.X.XXX.XXXXXX.XXXX.XXXXXXXXXXXXXXX.XXXXXXXXXXX.XXXX
(0020, 0010) Study ID SH: 'XXXXXXXXXXX'
(0020, 0011) Series Number IS: None
(0020, 0012) Acquisition Number IS: '1'
(0020, 0013) Instance Number IS: '1'
(0020, 0020) Patient Orientation CS: ''
(0020, 4000) Image Comments LT: ''
(0028, 0002) Samples per Pixel US: 3
(0028, 0004) Photometric Interpretation CS: 'RGB'
(0028, 0006) Planar Configuration US: 0
(0028, 0010) Rows US: 1024
(0028, 0011) Columns US: 1280
(0028, 0100) Bits Allocated US: 8
(0028, 0101) Bits Stored US: 8
(0028, 0102) High Bit US: 7
(0028, 0103) Pixel Representation US: 0
(0028, 2114) Lossy Image Compression Method CS: ''
(0040, 0244) Performed Procedure Step Start Date DA: ''
(0040, 0245) Performed Procedure Step Start Time TM: ''
(0040, 0253) Performed Procedure Step ID SH: 'XXXXXXXXXXX'
(0040, 0254) Performed Procedure Step Descriptio LO: ''
(0040, 0275) Request Attributes Sequence 1 item(s) ----
(0040, 0007) Scheduled Procedure Step Descriptio LO: ''
(0040, 0008) Scheduled Protocol Code Sequence 0 item(s) ----
(0040, 0009) Scheduled Procedure Step ID SH: 'XXXXXXXXXXX'
(0040, 1001) Requested Procedure ID SH: 'XXXXXXXXXXX'
---------
(0040, 0555) Acquisition Context Sequence 0 item(s) ----
(7fe0, 0010) Pixel Data OB: Array of 3932160 elements
.\ES00002.dcm
...画像表示してみる
matplotlibを使えば簡単。
import matplotlib.pyplot as plt
from pydicom import dcmread
from pydicom.data import get_testdata_file
import numpy as np
filename="./ES00001.dcm"
ds = dcmread(filename)
print(ds)
arr = ds.pixel_array
plt.imshow(arr, cmap="gray")
plt.show()
jpeg画像に変換する
これもPILで簡単。これで通常のjpeg画像として取り出すことができた。
from pydicom import dcmread
from PIL import Image
import glob
import os
output_dir = "jpg/"
if not os.path.exists(output_dir):
os.mkdir(output_dir)
l = glob.glob("./*.dcm")
for path in l:
#print(path)
ds = dcmread(path)
filename = os.path.splitext(path)[0]
arr = ds.pixel_array
im = Image.fromarray(arr)
im.save(output_dir + filename + ".jpg")
これで簡単に医用画像を一般的な画像データに変換することができた。
自分の場合は、過去の検査結果などをgoogle driveに保存していたりするので、同様に入れておき、何かあった時にかかりつけの医者にぱっとスマホで見てもらうこともできる。
これからの時代、それぞれの病院だけでデータを保持しているのは効率が悪いので、それが他の病院や本人が参照できるような時代になって欲しい。
それまでは自分で散財している自分の医療情報を把握するのはメリットがあるはず。


コメント